CIENCIAS BIOLÓGICAS Y DE LA SALUD
Un innovador enfoque brinda datos clave sobre la evolución de nuevos mecanismos de resistencia a antibióticos
Investigadores del CONICET estudian la estabilidad de las proteínas MBL en el contexto de la célula viva y responden interrogantes que podrían impactar en terapias clínicas para infecciones de microorganismos multirresistentes.
El grupo de investigación en Metaloproteínas del Instituto de Biología Molecular y Celular de Rosario (IBR, CONICET-UNR) publicó recientemente en la prestigiosa revista Nature Chemical Biology los resultados de un trabajo que aporta datos inéditos sobre la cinética y estabilidad dentro de la célula bacteriana de las Metalo-beta-lactamasas (MBLs), enzimas clave para conferir resistencia a los antibióticos de última generación. “En esta década hubo un gran aumento de las bacterias que tienen este mecanismo de resistencia. Su diseminación es muy preocupante, porque no hay manera de tratar las infecciones provocadas por estos microorganismos”, alerta Alejandro Vila, investigador del Consejo y director del laboratorio en IBR. Con el afán de aportar conocimientos que resulten útiles a esta problemática, diseñaron una aproximación experimental que integra el análisis bioquímico de las propiedades enzimáticas y la interacción con el contexto celular; esto les permitió identificar “el Talón de Aquiles de esta enzima en el ciclo de vida de la bacteria, que podría ser un blanco de ataque para desestabilizarla”, inactivando el mecanismo de resistencia bacteriano.
Evolución para resistir
¿Por qué aparecen cada vez más bacterias resistentes a los antibióticos? Primero hay que tener en cuenta que como las bacterias son organismos que están formados por una sola célula, su ciclo de vida dura lo que demora una célula en dividirse en dos nuevas células hijas, lo que suelen ser unos escasos treinta minutos. Esto hace que las bacterias tengan a su favor los tiempos evolutivos, pues en cada división celular ocurren modificaciones leves en su información genética. Algunos de estos cambios pueden resultar beneficiosos y las bacterias que los porten tendrán una ventaja adaptativa sobre el resto.
Segundo, se debe saber que los antibióticos se usan para detener el proceso de infección de las bacterias patógenas porque son moléculas que interfieren de modo específico con algún componente esencial en la vida de las bacterias, impidiendo su proliferación. Entonces, cuanto más se enfrenta las bacterias a los antibióticos, se aumenta la presión de selección y se van imponiendo las que llevan modificaciones genéticas que les permiten eludirlos. Esto acelera su evolución y favorece la transmisión de los genes de resistencia entre bacterias.
Es por esto que el uso excesivo e inadecuado de los antibióticos ha forjado un aumento de los casos clínicos con bacterias multirresistentes. De acuerdo con Vila, en particular, resulta preocupante el aumento de las que llevan genes para producir metalo-beta-lactamasas. Estas enzimas les permiten inactivar incluso a los antibióticos más potentes, los carbapenemes de última generación, y por ello representan un problema para la salud pública a nivel mundial.
El monitoreo de las infecciones producidas por la bacteria intra hospitalaria Klebsiella pneumoniae, realizado por el Servicio Antimicrobianos del Instituto Malbrán, ilustra la gravedad de la situación: el 30% de cepas encontradas en el país portan MBLs y son resistentes a carbapenemes. “En cierto sentido estaríamos volviendo a la era pre-antibiótica, se estima que para el año 2050 las infecciones bacterianas resistentes a antibióticos serían la primera causa de muerte en el mundo”, previene Lisandro González, investigador de CONICET y primer autor de trabajo.
El sistema contra ataca… y las bacterias también
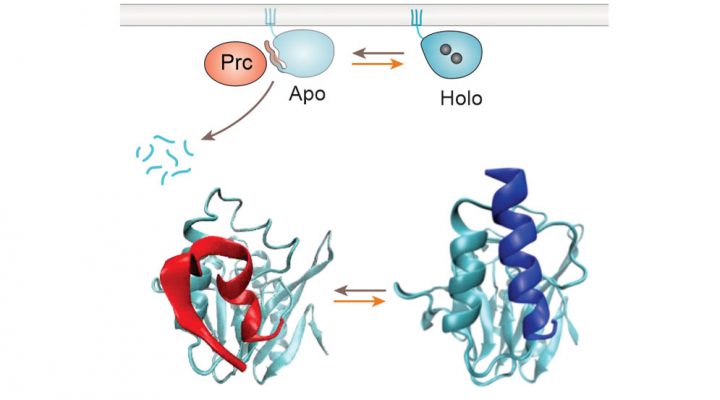
Ante la infección por un patógeno, el organismo humano reacciona inmediatamente para tratar de eliminarlo con las respuestas del sistema inmune innato. Según explica González, una de sus estrategias es remover los metales del medio donde se encuentran las bacterias. De esta forma inhiben el funcionamiento de muchas enzimas bacterianas que requieren metales en su estructura para funcionar correctamente. Las MBLs, como su nombre indica, llevan un metal (usualmente zinc) y también son afectadas por esta respuesta innata.
Utilizando un diseño experimental que combina técnicas de microbiología, microscopía y Resonancia Magnética Nuclear, los investigadores pudieron realizar un análisis bioquímico de la estabilidad de las variantes de MBLs de mayor relevancia clínica, pero en el contexto de la célula viva en condiciones de infección.
“Vimos que, ante la limitación de metales en el medio, la enzima queda frágil y es destruida por proteasas de la pared celular bacteriana”, detalla González y reconoce que recurrentemente aparecen en la clínica bacterias que llevan modificaciones en las enzimas MBLs, pero que los enfoques bioquímicos tradicionales in vitro no pueden explicar qué efecto tienen, por qué resultan beneficiosos y se seleccionan. En cambio, con estos experimentos pudieron demostrar que “el sistema inmune innato ejerce una fuerte presión que hace que se vayan seleccionando las bacterias que portan variantes de la enzima que resisten un poco más la falta de metal y eluden la degradación. Esta evolución está ocurriendo en las bacterias ahora”, explica.
A nivel especifico, probaron que para la estabilidad de las MBLs no solo es determinante su estado de unión al zinc sino también cómo logran ubicarse en un lugar particular de la pared de la célula bacteriana, donde quedan al resguardo de la acción de las proteasas.
De la mesada a la clínica
“Ahora que entendimos lo básico sobre la estabilidad de esta enzima, nos gustaría sacarle provecho para una aplicación clínica”, devela González y se pregunta si podrían encontrar compuestos que ayuden a desestabilizar a las MBLs, favoreciendo que las proteasas las ataquen.
Vila adelanta que están interesados en estudiar este sistema en bacterias patógenas como Klebsiella pneumoniae o Acetinetobacter baumannii, “porque probablemente haya que tener un abordaje distinto dependiendo de la bacteria y eso usualmente no se tiene en cuenta en la clínica”.
Por otro lado, González destaca su motivación por armar sistemas de estudio más realistas: “Sería valioso sumar más actores y trabajar con las bacterias junto con los glóbulos blancos que promueven la respuesta inmune innata, ya que es la defensa que se activa los primeros días y determina la gravedad de la infección”.
La ruta de las ideas nuevas
“Tratamos de unir los datos in vitro con lo que le pasa a la molécula en el contexto celular, en el marco de una pregunta que es relevante y tiene impacto en el campo de resistencia microbiana”, explica Vila. Para ello, destaca González: “Fue necesario pensar las cosas de otra manera, de ser creativos al integrar técnicas que estaban a nuestro alcance”.
Trabajar con la bioquímica enzimática dentro de la célula es un desafío, porque “hay tantas variables que abruman y no podés explorarlo todo”, reconoce González y cuenta que lo que más tiempo lleva en un trabajo como este es interpretar los resultados y pensar por donde seguir para acercarse a las respuestas.
Por su parte, Vila destaca que esta publicación tiene un alcance de gran escala porque brinda “un mapa de ruta para estudiar la estabilidad de las proteínas en el contexto celular” que podrían aplicarse a diversas temáticas biológicas. “Nos da mucho orgullo que una investigación que hacemos desde CONICET, en Rosario, tenga tanta repercusión y llegue a una revista tan importante. Está buenísimo reafirmar que estamos al mismo nivel que otros laboratorios del mundo”, valora con emoción González.
Por Elizabeth Karayekov – Área de Comunicación IBR (CONICET-UNR)