DIVULGACION CIENTIFICA
Gollum: una nueva herramienta para estudiar imágenes biológicas en super resolución
Investigadores del CIBION, del INIMEC y del IBioBA desarrollaron un software de código abierto que procesa y analiza imágenes de nanoscopía de fluorescencia para entender como funcionan las estructuras celulares. Esta herramienta es gratuita para toda la comunidad.
Una foto es como un corte transversal: muestra lo que pasa en un determinado espacio, en un determinado momento. Una serie de fotos es, al contrario, como un corte longitudinal y muestra – en el tiempo o en el espacio – los cambios que ocurren en determinadas estructuras.
Algo similar ocurre cuando se analizan las estructuras de las células con técnicas de nanoscopía de fluorescencia, que permiten ver estructuras de hasta 20 nanómetros (una mil millonésima parte de un metro). Si de esa estructura tomamos una foto, podemos determinar qué hay en ese espacio, en ese momento, dentro de la célula. Pero una serie de fotos, tomadas en distintos momentos, puede mostrar no sólo un panorama más completo sino que además permite reconstruir – en algunos casos – la función de la estructura que se analiza.
Para esto hace falta poder analizar y procesar automáticamente grandes cantidades – cientos, miles – de fotos. Un trabajo conjunto de investigadores del CONICET en el Centro de Investigaciones en Bionanociencias (CIBION), en el Instituto de Investigación Médica Mercedes y Martín Ferreyra (INIMEC, CONICET – IMMF – UNC) de Córdoba y en el Instituto de Investigación en Biomedicina de Buenos Aires (IBioBA – CONICET – Instituto Partner de la Sociedad Max Planck), permitió el desarrollo de un software de código abierto para cuantificar y procesar estructuras proteicas periódicas, es decir que se repiten. El trabajo fue publicado en la revista Scientific Reports.
“Creamos esta herramienta para poder analizar estructuras biológicas: una cosa es poder descubrirlas y saber que existen, pero es más difícil entender cómo funcionan. Para eso hay que sacar miles de imágenes bajo diferentes condiciones, y analizarlas, y entonces son necesarias herramientas de análisis automatizado”, explica Fernando Stefani, investigador principal del CONICET en el CIBION y coordinador del trabajo.
El proceso podría compararse con el descubrimiento de una nueva especie animal: si se le saca una foto ya se sabe que existe. Pero si se quiere saber cómo vive, se mueve o se reproduce hay que pasar varios años observándolo y tomando notas e fotos. Este nuevo software hace justamente eso: analiza gran cantidad de imágenes y cuantifica la cantidad y calidad de estructuras repetitivas dentro de la célula.
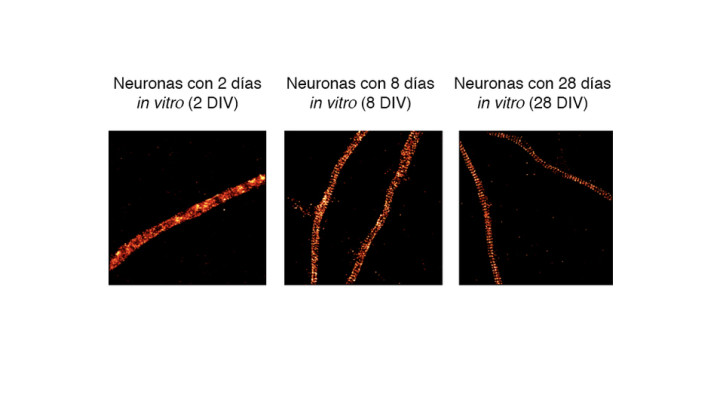
Para desarrollarlo, los grupos trabajaron con cultivos de neuronas de hipocampo donde estudiaron cómo se forman unos ‘anillos’ que están dentro de las prolongaciones de las neuronas (axones y dendritas) y que están formados por las proteínas actina y espectrina. Estas estructuras – que no se pueden observar con las técnicas clásicas – fueron descubiertas por microscopía de super resolución. Aparecen repetidamente dentro de las prolongaciones, están espaciadas cada 190 nanómetros y forman parte de su esqueleto interno. Como el software fue desarrollado para estudiar estos anillos, los investigadores lo llamaron Gollum en honor al personaje de El Señor de los Anillos.
“Sabíamos que aparecen en número variable en axones y dendritas. Por eso generamos muestras de neuronas, las observamos con microscopía de super resolución y las analizamos con este software en diferentes etapas de su crecimiento”, agrega Damián Refojo, investigador independiente del CONICET en el IBioBA, donde dirige el grupo de Neurobiología Molecular.
Así, vieron que este esqueleto regular se va estableciendo a medida que pasa el tiempo. Al principio, las neuronas inmaduras no lo tienen, pero al poco tiempo comienza aparecer e, in vitro, se termina de establecer entre los días 8 y 12 de vida.
“También notamos que la calidad del patrón aumenta: al comienzo es poco abundante e irregular, y después eso se va revirtiendo. Además vimos que no están a lo largo de todo el axón o dendrita, sino sólo en cerca del 50 o 60 por ciento de la longitud, lo que podría indicar que es una estructura dinámica, que se ensambla y desensambla activamente según las necesidades de la célula”, cuenta Alfredo Cáceres, investigador superior del CONICET en el INIMEC.
En este trabajo los autores presentan los algoritmos de análisis y un paquete de software que se puede aplicar al estudio de cualquier sistema biológico donde se quieran analizar estructuras periódicas del orden de los 20 nanómetros, que es el límite de resolución de los nanoscopios de fluorescencia.
“Hoy en día”, indica Refojo, “la nanoscopía de fluorescencia se está convirtiendo en el estándar para visualizar muestras biológicas y es fundamental contar con programas y aplicaciones que permitan analizar toda la información que pueden generar. Las posibilidades son casi infinitas”.
Acceso libre
El software que desarrolló el equipo de Stefani es parte de un esfuerzo para difundir el uso de nanoscopías de fluorescencia en la Argentina. Está disponible en un repositorio público y es de código abierto para que cada grupo pueda adaptarlo a sus necesidades. Esto permite – también – comparar y complementar datos analizados en diferentes laboratorios.
Los microscopios del CIBION están abiertos a la comunidad científica a través del Sistema Nacional de Microscopía, dependiente del Ministerio de Ciencia, Tecnología e Innovación Productiva de la Nación. “La propuesta es que la gente venga, aprenda y use las metodologías de nanoscopía y, si les sirve, que se puedan llevar el diseño de los nanoscopios, el software para controlarlo y las herramientas para analizar las imágenes”, concluye Stefani.
Acceso al software https://github.com/cibion-conicet/Gollum
Por Ana Belluscio. IBioBA – CONICET – MPSP.
Sobre investigación.
- Federico Barabas. CIBION. Departamento de Física, Facultad de Ciencias Exactas y Naturales, UBA.
- Luciano Masullo. Becario doctoral del CONICET. CIBION. Departamento de Física, Facultad de Ciencias Exactas y Naturales, UBA.
- Martín Bordenave. Departamento de Física, Facultad de Ciencias Exactas y Naturales, UBA.
- Sebastián Giusti. Investigador adjunto. IBioBA – CONICET – MPSP.
- Nicolás Unsain. Investigador asistente del CONICET. INIMEC (CONICET – IMMF – UNC). Universidad Nacional de Córdoba (UNC).
- Damián Refojo. Investigador independiente del CONICET. IBioBA – CONICET – MPSP.
- Alfredo Cáceres. Investigador superior del CONICET. INIMEC (CONICET – IMMF – UNC). Universidad Nacional de Córdoba (UNC).
- Fernando Stefani. Investigador principal del CONICET. CIBION. Departamento de Física, Facultad de Ciencias Exactas y Naturales, UBA.