CIENCIA CON VOZ PROPIA
La complejidad cabe en un gen
Descripto por primera vez hace más de 36 años, el proceso de splicing alternativo fascina a los científicos, ya que permite comprender como un puñado de genes pueden codificar para miles de proteínas.
Investigadores de CONICET publicaron en marzo un artículo de divulgación en la reconocida revista Nature Reviews in Molecular Cell Biology, donde quedó segundo en el ranking de los diez reviews más descargados en las últimas semanas. Y es que, entre otros temas, la regulación de este proceso puede explicar, al menos parcialmente, las diferencias evolutivas entre humanos y otros organismos como gusanos.
*Por Manuel J. Muñoz
El 15 y 16 de febrero de 2001 las revistas Nature y Science publicaron la secuencia y un primer análisis del genoma humano. Uno de los puntos más interesantes es que esos trabajos demostraron la baja cantidad de genes en nuestro genoma en relación a lo esperado. Como los genes en general codifican para proteínas, y su número es varias veces menor al de las proteínas producidas por el organismo, conocer los mecanismos que usa la célula para amplificar la información genética cobró vital importancia.
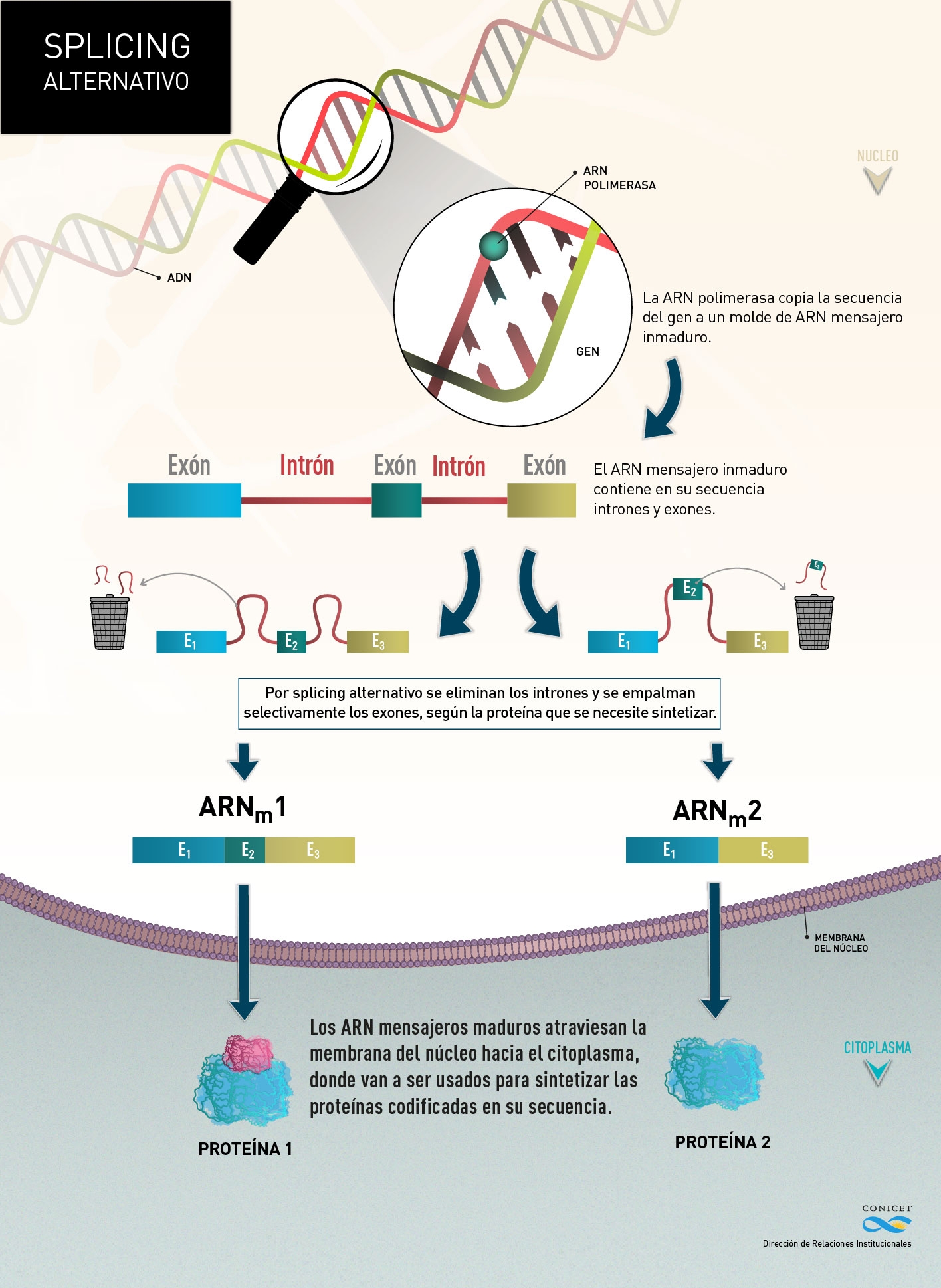
Este fue el caso del splicing alternativo, mecanismo por el cual más de un ARN mensajero – el ‘molde’ usado para fabricar proteínas – puede obtenerse a partir de un único gen.
(Click en la imagen para agrandar)
La expresión de un gen es un proceso complejo, tanto que obligó a los científicos a dividirlo en mecanismos más sencillos y acotados para analizarlos en profundidad. Sin embargo, décadas de estudio nos mostraron que es imposible seguir avanzando en la compresión de cómo funciona una célula sin entender la interconexión entre todos los pasos involucrados en la expresión de un gen.
Uno de los ejemplos más estudiados sobre cómo los procesos básicos en la expresión de un gen están conectados y se influencian entre sí es la regulación del splicing alternativo por la transcripción, el proceso en el que la secuencia de ADN de un gen se usa para fabricar una hebra de ARN mensajero, que luego será usado como molde para sintetizar proteínas.
Se encontró que la velocidad con la que se transcribe un gen afecta el splicing de ese mismo ARN mensajero. Mientras que la enzima que media este proceso, llamada ARN polimerasa, transcribe el ADN a ARN mensajero inmaduro, los llamados sitios de splicing o de unión entre intrones (secuencias que se removerán del ARN mensajero maduro) y exones (secuencias que sí se conservarán) son reconocidos por una maquinaria compleja llamada spliceosoma, que marca los ‘puntos de corte y pegado’ para el splicing o empalme selectivo.
Acá viene el dato: la arquitectura típica de un gen – un exón seguido de un intrón, este a su vez de otro exón y así sucesivamente – permite la competencia entre sitios de splicing por el spliceosoma. En caso de que el ARN sea sintetizado lentamente, un sitio de splicing débil (llamado así porque es débilmente reconocido por el spliceosoma) tendrá más tiempo para ser reconocido antes que el siguiente sitio de splicing, uno fuerte en este caso, sea sintetizado. En pocas palabras, no hay competencia si no hay con quien competir.
La clave es que el ARN mensajero maduro, que se obtiene tras el ‘corte y empalme’ del inmaduro, no será el mismo si ambos sitios – el fuerte, el débil o sólo uno – son reconocidos por el spliceosoma. Por lo tanto, el reconocimiento alternativo de sitios de splicing en el ARN inmaduro es la clave capaz de explicar cómo más de un ARN mensajero maduro es sintetizado a partir de un único gen.
En definitiva, la transcripción afecta el splicing: si la transcripción ocurre lentamente la probabilidad de que un sitio de splicing débil sea reconocido es mayor que si ocurre rápidamente. En ambas situaciones, transcripción rápida y transcripción lenta, los ARN mensajeros sintetizados no serán los mismos por lo que la velocidad con la que se sintetiza el ARN impacta sobre cuál ARN maduro es sintetizado.
La modulación de la velocidad transcripcional es sólo una de las tantas maneras por las cuales la maquinaria de transcripción es capaz de afectar decisiones de splicing alternativo pero en todos los casos el concepto relevante es el mismo: los sitios débiles dan opciones, sencillamente ser reconocidos o no, mientras que los sitios fuertes no las dan, son siempre reconocidos.
Por ejemplo nosotros, los humanos, tenemos un alto número de sitios de splicing débiles, por lo que muchos exones son alternativos y nuestra capacidad de amplificar la información genética es alta, mientras que en organismos más sencillos pero con una cantidad de genes similar, como las moscas o los gusanos, los exones alternativos son muchos menos y también lo son sus capacidades.
Haga click AQUÍ para acceder al review original en Nature Reviews – Mollecular cell Biology.
*Manuel J. Muñoz es investigador asistente del CONICET y trabaja en el Instituto de Fisiología, Biología Molecular y Neurociencias (IFIBYNE, UBA-CONICET). Obtuvo su licenciatura en Ciencias Biológicas en la UBA, y su doctorado en la misma universidad, con el tema “Respuesta al daño al ADN: un ejemplo de acoplamiento entre transcripción y splicing alternativo”.